資訊專欄INFORMATION COLUMN

本文主要是給大家介紹了pythonPyVCF文檔處理VCF文件類型范例詳細(xì)說明,感興趣的小伙伴值得借鑒參考一下,希望可以有一定的幫助,祝愿大家多多的不斷進(jìn)步,盡快工作上得到晉升
前言
vcf文件的全名是是variantcallfile,即突變性鑒別文檔,這是基因工作內(nèi)容過程中產(chǎn)生的一類文檔,存放的是基因里的突變性信息內(nèi)容。根據(jù)對vcf文件展開分析,可以獲得自我的基因變異信息內(nèi)容。嗯,總而言之,這是非常重要的文檔,因此如何處理它也變得極為重要。它文件信息如下:
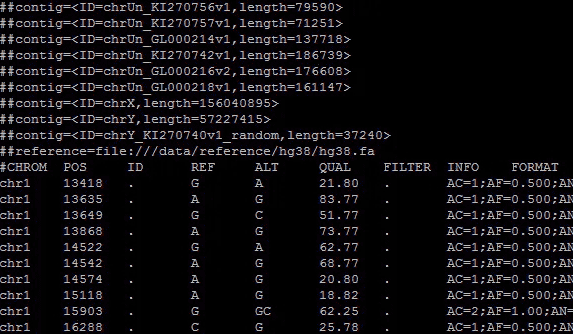
文檔開頭是一堆以“##”開始注解行,包括了文檔的相關(guān)信息。然后就是以“#”開頭一列,共9+若干個(gè)一部分,前九一部分注明的是后邊行每一部分意味著的數(shù)據(jù),等同于頁眉。后邊部分是樣本名稱,可以有多個(gè)。注解行完成后是實(shí)際的突變性信息內(nèi)容,每一行分成9+若干個(gè)一部分,每一部分間用分隔符(‘t’)隔開。
一般解決vcf文件時(shí),在載入,解決環(huán)節(jié)總會(huì)寫許多重復(fù)代碼,關(guān)鍵任務(wù)編碼非常少。自然,如果只是找結(jié)構(gòu)域的CHROM,POS,ID,REF,ALT,QUAL這些主要參數(shù)時(shí),這么做還可以。由于vcf格式標(biāo)準(zhǔn),這些參數(shù)構(gòu)造較為簡單。但如果解決庫函數(shù)信息內(nèi)容,或是解決INFO,F(xiàn)ORMAT主要參數(shù)時(shí),寫較為復(fù)雜的正則匹配,這么做不但繁雜,還很容易出差錯(cuò)。
Python的PyVCF庫克服了這種情況,主要是通過正則匹配把vcf文件信息內(nèi)容轉(zhuǎn)化成結(jié)構(gòu)型的數(shù)據(jù),優(yōu)化了vcf文件的處理方式,便捷后面獲取主要參數(shù)及解決。
PyVCF庫安裝
cmd界面
pipinstallPyVCF
或者從https://github.com/jamescasbon/PyVCF網(wǎng)站上下載安裝包,自行安裝。
PyVCF庫的導(dǎo)入
importvcf
PyVCF庫詳細(xì)介紹
使用范例:
>>>importvcf >>>vcf_reader=vcf.Reader(filename=r'D:testexample.hc.vcf.gz') >>>forrecordinvcf_reader: printrecordRecord(CHROM=chr1,POS=10146,REF=AC,ALT=[A])Record(CHROM=chr1,POS=10347,REF=AACCCT,ALT=[A])Record(CHROM=chr1,POS=10439,REF=AC,ALT=[A])Record(CHROM=chr1,POS=10492,REF=C,ALT=[T])Record(CHROM=chr1,POS=10583,REF=G,ALT=[A])
調(diào)用vcf.Reader類解決vcf文件,vcf文件信息內(nèi)容就被保存到vcf_reader中了。它是一個(gè)可迭代對象,它迭代元素都是一個(gè)_Record對象的范例,保存著非注解行的一列信息內(nèi)容,即基因變異結(jié)構(gòu)域的具體信息。通過它,我們可以很輕易地得到結(jié)構(gòu)域的詳細(xì)信息。
_Record對象------結(jié)構(gòu)域信息的儲(chǔ)存形式
classvcf.model._Record(CHROM,POS,ID,REF,ALT,QUAL,FILTER,INFO,FORMAT,sample_indexes,samples=None)
_Record是vcf.model中的一個(gè)對象,除了它還有_Call,_AltRecord等對象。它基本屬性為CHROM,POS,ID,REF,ALT,QUAL,F(xiàn)ILTER,INFO,F(xiàn)ORMAT,也就是vcf中的一列結(jié)構(gòu)域信息內(nèi)容。接下來對這些屬性一一說明:
CHROM:染色體名稱,類型為str。
POS:結(jié)構(gòu)域在染色體里的位置,種類為int。
ID:一般是突變性的rs號(hào),類型為str。如果是.’,則為None。
REF:參考基因組在這個(gè)結(jié)構(gòu)域里的堿基,類型為str。
ALT:在這個(gè)結(jié)構(gòu)域的測序結(jié)果。是_AltRecord類的派生類范例的目錄。種類為list。_AltRecord類有4個(gè)子類,代表著突變幾類種類:如snp,indel,structualvariants等。每一個(gè)范例都可以直接相對比較(僅限相等相對比較,并沒有大于小于之說),一部分派生類沒有實(shí)現(xiàn)str方法,也就是說不能轉(zhuǎn)成字符串。
QUAL:該結(jié)構(gòu)域的測序質(zhì)量,種類為int或float。
FILTER:過濾信息。將FILTER列按分號(hào)隔開形成的字符串目錄,種類為list。如果未給出主要參數(shù)則為None。
INFO:該結(jié)構(gòu)域的一些測試指標(biāo)。將‘=’前的主要參數(shù)作為鍵,后面的主要參數(shù)作為值,構(gòu)建成的字典。種類為dict。
FORMAT:基因型信息內(nèi)容。保存vcf的FORMAT列的原始形式
>>>for record in vcf_reader:
print type(record.CHROM),record.CHROM
print type(record.POS),record.POS
print type(record.ID),record.ID
print type(record.REF),record.REF
print type(record.ALT),record.ALT
print type(record.QUAL),record.QUAL
print type(record.FILTER),record.FILTER
print type(record.INFO),record.INFO
print type(record.INFO['BaseQRankSum']),record.INFO['BaseQRankSum']
print type(record.FORMAT),record.FORMAT
<type'str'>chr1<type'int'>
234481<type'NoneType'>None<type'str'>
T<type'list'>[A]<type'float'>2025.77<type'NoneType'>
None<type'dict'>{'ExcessHet':3.0103,'AC':[1],
'BaseQRankSum':-2.743,'MLEAF':[0.5],'AF':[0.5],
'MLEAC':[1],'AN':2,'FS':2.371,'MQ':42.83,
'ClippingRankSum':0.0,'SOR':0.972,'MQRankSum':-2.408,
'ReadPosRankSum':1.39,'DP':156,'QD':13.07}<type'float'>
-2.743<type'str'>GT:AD:DP:GQ:PL除了這些基本屬性之外,_Record對象還有一些其他屬性:
samples:把FORMAT信息作為鍵,后面對應(yīng)的信息做為值,構(gòu)建成的字典(CallData對象),以及sample名稱,這兩個(gè)值組成一個(gè)Call對象,共同構(gòu)成samples的一個(gè)元素。這樣就把sample和基因型信息給關(guān)聯(lián)起來,按下標(biāo)訪問每一個(gè)Call對象。samples類型為list。
start:突變開始的位置
end:突變結(jié)束的位置
alleles:該位點(diǎn)所有的可能情況,由REF和ALT參數(shù)組成的列表(REF類型是str,ALT參數(shù)是_AltRecord對象的子類實(shí)例),類型是list。
>>>for record in vcf_reader: print record.samples, 'n',record.samples[0].sample, 'n',record.samples[0]['GT'] #按下標(biāo)訪問Call,按.sample訪問sample,按鍵訪問FORMAT對應(yīng)信息 print record.start,record.POS,record.end print record.REF,record.ALT,record.alleles #注意G沒有引號(hào),它是_AltRecord對象 [Call(sample=192.168.1.1,CallData(GT=0/1,AD=[39,14], DP=53,GQ=99,PGT=0|1,PID=13116_T_G,PL=[449,0,2224]))] 192.168.1.10/113115 13116 13116T[G]['T',G]
_Record對象方法:
對象之間比較大小方法:根據(jù)染色體名稱和位置信息比較。Python3只實(shí)現(xiàn)了‘=’和‘<’的比較。
迭代方法:對samples里的元素進(jìn)行迭代。
字符串方法:只返回CHROM,POS,REF,ALT四列信息。
genotype(name)方法,和samples按下標(biāo)訪問不同,這個(gè)方法提供按sample名稱進(jìn)行訪問的功能。
add_format(fmt),add_filter(flt),add_info(info,value=True):給相應(yīng)的屬性添加元素。
get_hom_refs():拿到samples中該位點(diǎn)未突變的所有sample,返回列表。
get_hom_alts():拿到samples中該位點(diǎn)100%突變的所有sample,返回列表。
get_hets():拿到samples中該位點(diǎn)基因型為雜合的所有sample,返回列表。
get_unknown():拿到samples中該位點(diǎn)基因型未知的所有sample,返回列表。
>>>record=next(vcf_reader)
>>>record2=next(vcf_reader)
>>>print record>record2
#按染色體名稱和位置進(jìn)行比較False
>>>for i in record:
#按samples列表進(jìn)行迭代
print i
Call(sample=192.168.1.1,
CallData(GT=0/1,AD=[18,11],
DP=29,GQ=99,PL=[280,0,528]))
>>>print str(record)
#字符串方法Record(CHROM=chr1,POS=10492,REF=C,ALT=[T])
>>>print record.genotype('192.168.1.1')
#按sample名字進(jìn)行訪問
Call(sample=192.168.1.1,
CallData(GT=0/1,AD=[39,14],DP=53,GQ=99,PGT=0|1,PID=13116_T_G,PL=[449,0,2224]))
_Record對象還有很多有用的方法屬性:
num_called:該位點(diǎn)已識(shí)別的sample數(shù)目。
call_rate:已識(shí)別的sample數(shù)目占sample總數(shù)的比例。
num_hom_ref,num_hom_alt,num_het,num_unknown:四種基因型的數(shù)量
aaf:所有sample等位基因的頻率(即除開REF),返回列表。
heterozygosity:該位點(diǎn)的雜合度,0.5為雜合突變,0為純合突變。
var_type:突變類型,包括‘snp’,‘indel’,‘sv’(structural variant),‘unknown’。
var_subtype:更加細(xì)化的突變類型,如‘indel’包括‘del’,‘ins’,‘unknown’。
is_snp,is_indel,is_sv,is_transition,is_deletion:判斷突變是不是snp,indel,sv,transition,deletion等等。
>>>record=next(vcf_reader)
>>>print recordRecord(CHROM=chr1,POS=13118,REF=A,ALT=[G])
>>>print record.samples
#只有一個(gè)sample
[Call(sample=192.168.1.1,CallData(GT=0/1,AD=[41,13],DP=54,GQ=99,PGT=0|1,PID=13116_T_G,PL=[449,0,2224]))]
>>>record.num_called1
>>>record.call_rate1.0
>>>record.num_hom_ref0
>>>record.aaf[0.5]
>>>record.num_het1
>>>record.heterozygosity0.5
>>>record.var_type'snp'
>>>record.var_subtype'ts'
>>>record.is_snpTrue
>>>record.is_indelFalseReader對象------處理vcf文件,構(gòu)建結(jié)構(gòu)化信息
class Reader(fsock=None,filename=None,compressed=None,prepend_chr=False,strict_whitespace=False,encoding='ascii')
在讀vcf文件時(shí),總共有六個(gè)參數(shù)可供選擇,如上圖所示。
fsock:目標(biāo)文件的文件對象,可以用open(文件名)得到這個(gè)文件對象。
filename:文件名,當(dāng)fsock和filename同時(shí)存在時(shí),優(yōu)先考慮fsock。
compressed:是否要解壓,不提供參數(shù)時(shí)由程序自行判斷(以文件名是否以.gz結(jié)尾判斷是否需要解壓)。
prepend_chr:在保存染色體名稱時(shí),是否加前綴‘chr’,默認(rèn)不加,如果vcf文件的染色體名稱本來沒有前綴‘chr’,可設(shè)置為True,自動(dòng)加上。
strict_whitespace:是否嚴(yán)格以制表符‘t’分隔數(shù)據(jù)。True則表示嚴(yán)格按制表符分,F(xiàn)alse表示可以夾雜空格。
encoding:文件編碼。
>>>vcf_reader=vcf.Reader(open('vcf/test/example-4.0.vcf','r'))
#fsock
>>>vcf_reader=vcf.Reader(filename=r'D:testexample.hc.vcf.gz')
#filename頭文件信息主要保存在Reader對象的屬性中,包括alts,contigs,filters,formats,infos,metadata。
alts使用實(shí)例:
>>>vcf_reader=vcf.Reader(filename=r'D:testexample.hc.vcf.gz')
>>>vcf_reader.altsOrderedDict([('NON_REF',Alt(id='NON_REF',desc='Represents any possible alternative allele at this location'))])#字典類型
>>>vcf_reader.alts['NON_REF'].id'NON_REF' >>>vcf_reader.alts['NON_REF'].desc'Represents any possible alternative allele at this location'
其他的屬性用法類似。
Reader對象實(shí)現(xiàn)了兩個(gè)方法:
next():獲得下一行的數(shù)據(jù),也就是返回下一個(gè)_Record對象。可以顯式調(diào)用next()得到下一行數(shù)據(jù),也可以直接迭代Reader對象,它會(huì)自動(dòng)調(diào)用next()函數(shù)以獲得下一行數(shù)據(jù)。
fetch(chrom,start=None,end=None):返回chrom染色體從start+1到end坐標(biāo)的所有突變位點(diǎn)。不給end,就返回chrom染色體從start+1到末尾的所有突變位點(diǎn);
start和end都不給,就返回chrom染色體所有的突變位點(diǎn)。這個(gè)方法需要用另一個(gè)第三方Python模塊pysam來建立文件索引,如果沒有安裝這個(gè)模塊,會(huì)導(dǎo)致錯(cuò)誤。
另外,使用這個(gè)方法之后,它會(huì)將對象的可迭代范圍改成fetch()得到的突變位點(diǎn),所以用這個(gè)方法,原來的迭代進(jìn)度就失效了。
>>>vcf_reader=vcf.Reader(filename=r'D:testexample.hc.vcf.gz') >>>vcf_reader.next()<vcf.model._Record object at 0x0000000003ED8780 >>>>record=vcf_reader.next() >>>print recordRecord(CHROM=chr1,POS=10347,REF=AACCCT,ALT=[A]) >>>for record in vcf_reader: print recordRecord(CHROM=chr1,POS=10439,REF=AC,ALT=[A])Record(CHROM=chr1,POS=10492,REF=C,ALT=[T])
這個(gè)庫還有一個(gè)Writer對象,在此就不詳細(xì)介紹了,因?yàn)榇蟛糠謱cf文件的處理都可以用上面兩個(gè)對象的知識(shí)搞定。
綜合使用:
#!/usr/bin/env python #-*-coding:utf-8-*-import vcf #導(dǎo)入PyVCF庫 filename=r'D:testexample.hc.vcf.gz' vcf_reader=vcf.Reader(filename=filename) #調(diào)用Reader對象處理vcf文件 for record in vcf_reader: #迭代Reader對象,返回的是_Record對象 #record是_Record對象 print record.CHROM,record.POS,record.ID,record.ALT if record.is_snp: #判斷是否是snp print"I'm a snp" elif record.var_type!='sv': #和elif record.is_sv:等價(jià) print"I'm not a sv" if record.heterozygosity==0.5: #判斷是否為雜合突變 print"I'm a heterozygous mutation" ......
這個(gè)庫實(shí)現(xiàn)的所有功能,都可以自己寫代碼實(shí)現(xiàn),而且實(shí)現(xiàn)方法比較簡單。之所以要用這個(gè)庫來處理vcf文件,是因?yàn)檫@個(gè)庫考慮的東西可能比我們自己了解的更多,其實(shí)現(xiàn)也可能比我們自己的代碼更加完備合理。
還有,重復(fù)造車總歸是不好的。
綜上所述,這篇內(nèi)容就給大家介紹到這里了,希望可以給各位讀者帶來幫助。
文章版權(quán)歸作者所有,未經(jīng)允許請勿轉(zhuǎn)載,若此文章存在違規(guī)行為,您可以聯(lián)系管理員刪除。
轉(zhuǎn)載請注明本文地址:http://m.specialneedsforspecialkids.com/yun/128446.html
小編寫這篇文章的主要目的,主要是給大家去做一個(gè)解答,解答的內(nèi)容,主要是實(shí)現(xiàn)GATK多線程加速,那么,具體的一個(gè)操作手法是什么呢?怎么去實(shí)現(xiàn)加速呢?下面就給大家詳細(xì)解答下。 GATK變異分析 對于大數(shù)據(jù)樣本可能會(huì)比較慢,因此可以按照染色體拆分后進(jìn)行多線程并行計(jì)算。 下面是我寫的一個(gè)python多線程腳本,僅供參考,拙劣之處敬請指正。 #!/usr/bin/python3 import...
摘要:一積累中如何快速查看包中的源碼最常用的大開發(fā)快捷鍵技巧將對象保存到文件中從文件中讀取對象中的用法的配置詳解和代碼的格式詳解格式化內(nèi)容設(shè)置生成詳解注釋規(guī)范中設(shè)置內(nèi)存調(diào)試的小知識(shí)單步執(zhí)行命令的區(qū)別的動(dòng)態(tài)代理機(jī)制詳解內(nèi)容有瑕疵,樓指正泛型繼承的幾 一、積累 1.JAVA Eclipse中如何快速查看jar包中 的class源碼 最常用的15大Eclipse開發(fā)快捷鍵技巧 Java將對象保存到...
摘要:一積累中如何快速查看包中的源碼最常用的大開發(fā)快捷鍵技巧將對象保存到文件中從文件中讀取對象中的用法的配置詳解和代碼的格式詳解格式化內(nèi)容設(shè)置生成詳解注釋規(guī)范中設(shè)置內(nèi)存調(diào)試的小知識(shí)單步執(zhí)行命令的區(qū)別的動(dòng)態(tài)代理機(jī)制詳解內(nèi)容有瑕疵,樓指正泛型繼承的幾 一、積累 1.JAVA Eclipse中如何快速查看jar包中 的class源碼 最常用的15大Eclipse開發(fā)快捷鍵技巧 Java將對象保存到...
摘要:什么是推導(dǎo)式大家好,今天為大家?guī)韱栁易钕矚g的推導(dǎo)式使用指南,讓我們先來看看定義推導(dǎo)式是的一種獨(dú)有特性,推導(dǎo)式是可以從一個(gè)數(shù)據(jù)序列構(gòu)建另一個(gè)新的數(shù)據(jù)序列的結(jié)構(gòu)體。 什么是推導(dǎo)式 大家好,今天為大家?guī)韱栁易钕矚g的Python推導(dǎo)式使用指南,讓我們先來看看定義~ 推導(dǎo)式(comprehensions)是Python的一種獨(dú)有特性,推導(dǎo)式是可以從一個(gè)數(shù)據(jù)序列構(gòu)建另一個(gè)新的數(shù)據(jù)序列的結(jié)構(gòu)體。...
閱讀 926·2023-01-14 11:38
閱讀 899·2023-01-14 11:04
閱讀 758·2023-01-14 10:48
閱讀 2063·2023-01-14 10:34
閱讀 965·2023-01-14 10:24
閱讀 844·2023-01-14 10:18
閱讀 512·2023-01-14 10:09
閱讀 590·2023-01-14 10:02